La leucemia mieloide aguda es una neoplasia heterogénea. Las características bioquímicas de tal enfermedad son múltiples y diversas. Las aberraciones cromosómicas aparecen frecuentemente. El gen NPM1 aparece alterado en el 50% de los pacientes, mientras que el FLT3 es aberrante en hasta el 40% aunque éste último generalmente es sinónimo de mal pronóstico; existen otros genes mutados, los que le confieren una tasa replicativa mayor o una pérdida de la diferenciación potencial. Ninguna mutación por sí sola es capaz de convertir a una célula en leucémica sino que se requieren cambios adyuvantes. La mayor parte de estos cambios se correlaciona con un pronóstico.
Revisión bibliográfica. Biología molecular de leucemia mieloide aguda
Dra. Bismay Machado Cobas (1), Lic. Alexei Santana Galano (2), Dra. Mirley Vilató Floriam (3), Dr. Orlando Sanamé Columbié. (4)
1 Máster en Atención Integral a la Mujer. Especialista de I Grado en Medicina
General Integral. Asistente.
2 Licenciado en Bioquímica. Instructor.
3 Máster en Atención Integral a la Mujer. Especialista de I Grado en Medicina General Integral. Instructor.
4 Especialista de I Grado en Anatomía Patológica. Instructor.
Entidad: Filial Ciencias Medicas Baracoa. Guantánamo. Cuba
RESUMEN
La leucemia mieloide aguda es una neoplasia heterogénea. Las características bioquímicas de tal enfermedad son múltiples y diversas. Las aberraciones cromosómicas aparecen frecuentemente. El gen NPM1 aparece alterado en el 50% de los pacientes, mientras que el FLT3 es aberrante en hasta el 40% aunque éste último generalmente es sinónimo de mal pronóstico; existen otros genes mutados, los que le confieren una tasa replicativa mayor o una pérdida de la diferenciación potencial. Ninguna mutación por sí sola es capaz de convertir a una célula en leucémica sino que se requieren cambios adyuvantes. La mayor parte de estos cambios se correlaciona con un pronóstico.
Palabras clave: leucemia mieloide aguda, cáncer, tumor hemolinfopoyético.
INTRODUCCIÓN
La leucemia mieloide aguda (LMA) es una enfermedad difícil de tratar; afecta fundamentalmente a los adultos entre 15 y 39 años y constituye el 20% de las leucemias de la infancia. La leucemia mieloide aguda (LMA) es extraordinariamente heterogénea, reflejando las complejidades de la diferenciación de las células mieloides (la leucemia mieloide aguda (LMA), según la clasificación FAB revisada, agrupa 8 tipos de leucemias –M0 a M7-, y varios subtipos).
La leucemia mieloide aguda (LMA) es un proceso con múltiples pasos que involucran al menos dos tipos de mutaciones, las de clase I que activan las vías de transducción de señales confiriendo ventajas de proliferación sobre las células hematopoyéticas, y las mutaciones de clase II que involucran factores de transcripción y repercuten sobre la diferenciación celular.
En el presente trabajo nos hemos propuesto como objetivo brindar los principales elementos moleculares conocidos sobre la etiología de dicha enfermedad.
DESARROLLO
ALTERACIONES CARIOTÍPICAS
Las alteraciones cariotípicas aparecen en el 90% de los pacientes con leucemia mieloide aguda (LMA), aunque con las técnicas de tinción habituales generalmente es posible visualizar la mitad. Se han identificado una serie de alteraciones cromosómicas en pacientes con leucemia mieloide aguda (LMA). Entre ellas cabe destacar monosomías de los cromosomas 5 y 7, deleciones del brazo largo del cromosoma 5, anormalidades en el brazo largo del cromosoma 3, y otras anormalidades complejas. Se ha definido un cariotipo monosómico (CM) como de alto riesgo, el que involucra dos o más monosomías, así como una monosomía y otra aberración estructural; así mismo, se han definido grupos de bajo riesgo y riesgo intermedio, atendiendo fundamentalmente a la agresividad y resistencia de la enfermedad al tratamiento. No obstante, la razón molecular del por qué de este aumento del riesgo, permanece generalmente oscura.
Las leucemias mieloides agudas (LMA) que surgen de novo en los pacientes sin ningún factor de riesgo conocido suelen cursar con translocaciones cromosómicas equilibradas, mientras que aquellas que aparecen luego de un contacto con algún agente lesivo al ADN (como quimioterapia o radiaciones) suelen asociarse con deleciones o monosomías (ejemplo: cromosomas 5 y 7), y generalmente sin translocaciones. Sin embargo, aquellos agentes terapéuticos que inhiben la topoisomerasa II se asocian a translocaciones en 11q23, en el gen MLL.
OTRAS ALTERACIONES
Aproximadamente el 50% de los pacientes no presentan alteraciones cariotípicas visibles por las técnicas de tinción convencionales. Sin embargo, cada vez son más los genes que se asocian a la aparición y evolución de las leucemias mieloides agudas (LMA).
NPM1
Las mutaciones en el gen de la nucleofosmina 1 (NPM1) constituyen las mutaciones más frecuentes en aquellos pacientes sin aberraciones cromosómicas visibles, apareciendo hasta en el 50% de los pacientes con cariotipo normal, más abundante en la variante M4 y en células carentes de CD34. Son particularmente frecuentes en el exón 12 de dicho gen, todas afectando el C-Terminal de la proteína, identificándose hasta la fecha 14 variantes (todos mutantes heterocigóticos), con inserción de generalmente 4 pares de bases (pb), lo que produce un corrimiento del marco de lectura, y la sustitución de los últimos 7 aminoácidos por 11 diferentes residuos (los últimos cinco aminoácidos VSLRK, son comunes a todas las variantes), así como la sustitución de los triptófanos 288 y 290.
La proteína se localiza normalmente en el nucleolo, y se explica la presencia anormal de NPM1 en citoplasma por la presencia de una secuencia señal (como resultado de las mutaciones) de exportación del núcleo. La mutación de NPM1 es más común en mujeres (sobre el 60%).
Las funciones de NPM1 incluyen:
a) Interactúa con p53 controlando la proliferación celular y la apoptosis.
b) Ayuda a mantener la estabilidad genómica controlando la reparación del ADN y la duplicación de los centrosomas durante la mitosis.
c) Controla la biosíntesis de ribosomas.
Lógicamente, mutantes defectuosos de NPM1 afectarán el ciclo celular. NPM1 forma parte de un nuevo grupo de genes que pueden actuar como oncogenes o genes supresores tumorales. Sin embargo, su expresión sin FLT3 favorece un buen pronóstico.
FLT3
Aproximadamente un tercio de los pacientes afectados por leucemia mieloide aguda (LMA) tienen mutaciones en el gen FLT3, pudiendo clasificarse esta mutación dentro de la clase I.
El gen FLT3 codifica para un receptor, perteneciente a la clase III de la familia de receptores tirosina quinasa; posee 993 aminoácidos (aa), y un peso molecular de158/160 kD, está glicosilado (la variante no glicosilada no se asocia a membrana y pesa 130/143 kD), su locus aparece en 13q12, poseyendo 24 exones. El ligando de FLT3 (FL) es una proteína de membrana de tipo 1, con 235 aa, repartidos en un péptido señal hacia el N Terminal, cuatro dominios helicoidales extracelulares, regiones espaciadoras y regiones bisagra, un dominio transmembrana y un corto dominio citoplasmático. FL aparece en muchos tejidos, incluyendo aquellos que componen órganos hematopoyéticos, con los niveles más altos de expresión en células mononucleares de sangre periférica.
La unión de FL a FLT3 provoca una autofosforilación del receptor en residuos de tirosina, creando sitios de unión para moléculas efectoras y diversas vías de señalización. El dominio citoplasmático de FLT3 se une a la subunidad p85 del fosfoinositol 3 quinasa (PI3K), RAS GTPasa, fosfolipasa C-γ, Shc, Grb2 y proteínas quinasas de la familia Src; como resultado, estas proteínas son fosforiladas; posteriormente resultan afectadas las vías que involucran PI3Kproteína quinasa B (Akt) y la proteína quinasa activada por mitógeno (MAPK) (Figura 1). Es de esperar que, dado el papel que desempeña FLT3 en la expresión de proteínas y en el ciclo celular, la afectación del receptor, tenga efectos fisiológicos profundos.
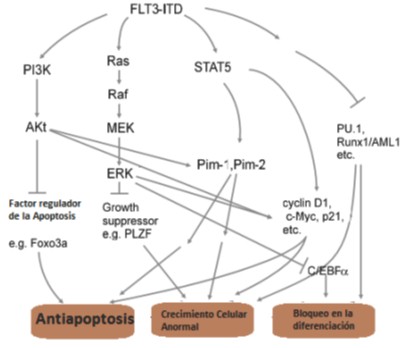
Figura 1. Mecanismos mediante los cuales FLT3 induce la oncogénesis leucémica. (Tomado de Takahashi S., Downstream molecular pathways of FLT3 in the pathogenesis of acute myeloid leukemia: biology and therapeutic implications. J. Hematol. Oncol. 2011, 4:13).
La mayor parte de las mutaciones involucran una duplicación en tándem del dominio yuxtamembrana. Los cambios en el receptor por si solos no son capaces de convertir una célula en tumoral, sino que colaboran en el “almacenamiento” de defectos oncológicos. De hecho, la mutación anteriormente descrita aparece en pacientes con mutaciones en la nucleofosmina y DNA metil transferasa 3A, y el 50% de pacientes con translocaciones t(15,17). Aun cuando la longitud del tándem repetido puede variar, FLT3 mantiene su orientación, pero se piensa que el cambio conformacional en el dominio yuxtamembrana provoca una dimerización y activación del receptor. Esta mutación aparece en el 25% de los pacientes. Se describen un 7% con cambios en el Asp 835.
Las mutaciones de FLT3 influyen ligeramente de forma negativa en el logro de remisiones completas en adultos; sin embargo, existe una correlación inversa entre la edad y la influencia nefasta de los mutantes a la hora de analizar el éxito terapéutico.
Las mutaciones de FLT3 activan de forma permanente el receptor, provocando que se pierda la capacidad de auto inhibición. Entre las tantas vías que se activan, de especial interés es la de STAT5, que activa la transcripción de genes blanco ciclina D1, c-myc, y el gen antiapoptótico p21, así como enzimas serina quinasas de la familia Pim (Pim 1 y Pim2).
Otro efecto fisiológico importante de la activación constitutiva de FLT3, igualmente, mediado por STAT5 es la sobreproducción de Especies Reactivas del Oxigeno (ERO), las que pueden dañar el ADN. La fosforilación de STAT5 favorece la actividad del complejo RAC1-GTP, lo que favorece la aparición de ERO a partir de la NADPH oxidasas. FLT3 activa Akt y este provoca una fosforilación en la treonina 32 de la proteína FOXO3a (perteneciente a la familia de proteínas Forkhead), translocándose está desde el núcleo al citoplasma, evitando que FOXO3a pueda desempeñar funciones apoptóticas. FLT3 activa la proteína quinasa regulada por señal extracelular (ERK), la que a su vez fosforila el residuo Ser21 del complejo C/EBP_ (factor de transcripción mieloide), lo que evita la diferenciación celular.
La proteína PLZF (pro-myelocytic leukemia zinc finger) es un represor transcripcional y un potente represor del crecimiento que bloquea la proliferación celular y la diferenciación mieloide, mediante el silenciamiento de genes como la ciclina A2. Se ha demostrado que los mutantes patológicos de FLT3 disocian la proteína PLZF, así como los receptores de la hormona tiroidea y del ácido retinoico (SMRT), provocando crecimientos aberrantes de la línea mieloide. Por otra parte, los receptores aberrantes de FLT3 son capaces de romper las interacciones efectivas entre SMRT y factores de transcripción (represores) como RunX1/AML1 (inductores de “vejez celular”), con una expresión concomitante y aberrante de p21WAF1/CIP1.
Se han desarrollado una serie de drogas que mimetizan las purinas en el sitio activo del FLT3, entre ellas SU11248 (sunitinib), MLN518 (tandutinib), CEP-701 (lestaurtinib) and PKC412 (midostaurin), las que están en fase clínica, aunque todo parece indicar que la respuesta es incompleta y de corta duración. AC220 parece tener mejores resultados.
CEBPA
El gen CEBPA (19q13.1) codifica para una proteína que se une a la secuencia CCAAT de promotores de algunos genes con funciones específicas en los granulocitos, un factor de diferenciación de granulocitos importante en la regulación de los precursores mieloides.
Las mutaciones en CEBPA (C/EBP_) aparecen en el 5-10% de todas las nuevas LMA, más frecuentemente en aquellas con cariotipo normal. El pronóstico generalmente se considera bueno, y es más común en M1 o M2.
La proteína C/EBP_ (de 42kD) aumenta su actividad por fosforilación mediada por RAS, de la Ser248 del dominio transactivador de la proteína. La proteína, hacia el C-Terminal, consta de motivos altamente homólogos de unión al ADN y para dimerización (zipper de Leucina); mientras que hacia el N-Terminal aparecen motivos menos conservados de transactivación;.pueden ser de varios tipos las mutaciones en C/EBP.
Mutaciones “sin sentido” hacia el N-Terminal producen fundamentalmente una proteína truncada de 20kDa, y una proteína de 30kDa por el uso de un codón alternativo de inicio en el aminoácido 120, utilizando el mismo marco de lectura (p30C/EBPα, dominante).
Aun cuando exista un alelo salvaje, las proteínas aberrantes son capaces de “enmascarar” los efectos de las “normales”. Hacia el C Terminal se describen fundamentalmente deleciones, inserciones o duplicaciones.
En pacientes con t(8;21)(q22;q22), la proteína quimérica AML1-ETO resultante reprime la transcripción de CEBPA a niveles insuficientes para la diferenciación normal de los granulocitos, evitando su unión al ADN y autorregulación. De igual manera, la proteína PML/RARα se une al CEBPA y el complejo resultante es incapaz de unirse al ADN.
La proteína CEBP_ interactúa con E2F, p21 y Cdk2Cdk4. La inhibición de la actividad de E2F por CEBP_ permite una expresión de c-Myc, siendo este paso crucial en la diferenciación granulocítica.
MLL
Las mutaciones en el gen MLL (Mixed-Lineage-Leukemia) (localizado en el locus 11q23) también aparecen en los pacientes LMA. Las mutaciones más frecuentes son t(6;11) (q27;23) (MLL-AF6), t(9;11) (p23;q23) (MLL-AF9), t(10;11) (p12;q23) (MLL-AF10) y t(11;19) (q23;13.1) (MLLELL).
El gen MLL contiene 36 exones, y codifica para una proteína de 3969 aminoácidos (y un peso molecular de 430 kDa), que funge como regulador positivo en el desarrollo embrionario temprano y la hematopoyesis. Este gen, tiene un punto de ruptura sobre la región de 8.3 kB (entre los exones 5-11), todas las proteínas quiméricas MLL mantienen la región amino terminal, la cual se une al ADN, mientras que el segmento restante pierde las funciones reguladoras sobre la actividad de la proteína, expresándose aberrantemente una serie de genes como los HOX, EPHA7, MEIS y PBX, los que influyen sobre la mayor tasa de aparición de células con potencial tumoral.
El pronóstico de la leucemia mieloide aguda (LMA) MLL-AF6 es extremadamente malo, con una tasa de supervivencia (TS) a los 5 años generalmente menor al 20% en todos los grupos de edades. Los asiáticos parecen tener peor pronóstico.
Para el caso de MLL-AF9 el pronóstico es intermedio o bueno, con la tasa de supervivencia (TS) sobre el 50%, aunque los resultados son variables entre los distintos grupos poblacionales.
Los japoneses tienen muy mal pronóstico para la MLL-ELL sobre el 10% de supervivencia al año, aunque a nivel mundial oscila sobre el 60%, con mejor pronóstico para los niños.
KIT
Las mutaciones en KIT aparecen entre el 30-40% de los pacientes con inv(16)(p13.q22) lo que se interpreta como un pronóstico peor. Las mutaciones KIT (particularmente en D816) donde se observen t(8;21)(q22;q22) aparecen entre el 20-30%, e igualmente el pronóstico es sombrío.
El receptor tirosina-quinasa codificado por el gen KIT, parece tener un mecanismo fisiopatológico similar al FLT3. Por tanto, determinadas mutaciones en KIT le confieren a los mieloblastos actividad proliferativa y |o antiapoptótica. Resulta intrigante la asociación de las mutaciones en KIT con inv16, t(8;21), y trisomía 4. Las mutaciones puntuales en KIT aparecen en el 5% de los nuevos casos LMA. La mutación más importante en KIT es D816Y/H, seguido de delecionesinserciones que afecten al exón 8. La trisomía 4 confiere mal pronóstico.
El gen AML1 (RUNX1) aparece involucrado en un número importante de translocaciones, especialmente en t(8;21), en el 12/15% de los pacientes con leucemia mieloide aguda (LMA). Así mismo, el gen de fusión CBFß /MYH11 producido por la yuxtaposición de las bandas 16q22 16p13 como resultado de inv16 o menos frecuentemente t(16;16)(p13;q22) esta proteína quimérica es capaz de bloquear la diferenciación de los mieloblastos solo en presencia de KIT.
p53
La mayoría de las neoplasias sólidas requieren para su desarrollo tumoral de la inactivación del gen p53. Sin embargo la literatura describe mutaciones inactivantes homocigóticas aproximadamente en el 6% de los pacientes con leucemia mieloide aguda (LMA). El gen de p53 se localiza en el 17p13.1, y en las leucemias mieloides agudas (LMA) se plantea la existencia de una “zona común” de alteraciones que se circunscribe entre las 6828482–8075871pb (aproximadamente 1.25 Megabases), donde además de p53 aparecen otros genes como CRK y HIC1.
La mutación más frecuente en p53 en el caso de las leucemias mieloides agudas (LMA) fueron las mutaciones “sin sentido” (missense, especialmente entre los residuos aminoacídicos 102 al 292), apareciendo un alto número de mutantes homocigóticos. Se piensa que en el primer paso en la pérdida de las funciones de p53, un alelo es inactivado por mutación, y el otro se pierde fundamentalmente por deleción (la presencia de dobles mutantes heterocigóticos es poco frecuente); los mutantes homocigóticos requerirían un tercer paso: la reduplicación del alelo mutante como un mecanismo de compensación cromosomita. Dada la importancia que tiene p53 en el control de la reparación al ADN, así como en el ciclo celular, no es de asombrarse que pacientes con ambas copias de p53 defectuosas, presenten además aberraciones en los cromosomas 5 y 7, y características histomorfológicas agresivas.
OTROS GENES
El producto de t(15;17)(q22;q12) es PML/RARα. El gen de fusión codifica un receptor anormal del ácido retinoico que impide la diferenciación de las células mieloides, probablemente porque interfiere el funcionamiento de otros receptores normales. Dosis elevadas de ácido retinoico vencen dicha competencia, y permiten la diferenciación y el tratamiento de las leucemias resultantes, aunque no eliminan el clon neoplásico.
AML1 es capaz de reclutar varias proteínas involucradas en la acetilación de histonas (p300), y activar una serie de genes necesarios en la maduración mieloide. En la t(8;21) AML1 se recombina con la proteína nuclear ETO, compitiendo AML1ETO con AML1 “salvaje”, inhibiendo la transcripción de una serie de genes.
Se han descrito mutaciones en los genes de la enzima Isocitrato Deshidrogenasa I (en el 10-15% de los nuevos casos de leucemia mieloide aguda (LMA)), con pronósticos adversos; de igual forma, ciertas mutaciones en Isocitrato Deshidrogenasa II (descritas recientemente) se correlacionan con fallos en la terapéutica. Mutaciones en el gen TET2 aparecen en el 25% de los pacientes con leucemia mieloide aguda (LMA) secundaria (aunque se desconoce el impacto). Mutantes MLL aparecen en el 5% de los pacientes, especialmente en las leucemias mieloides agudas (LMA) recurrentes, con pronóstico malo. Igualmente se han descrito casos de mutantes en BCR-ABL.
Algunos pacientes expresan el receptor de quimoquinas CXCR4 (contra el que hay anticuerpos) y la proteína P-gp, que es capaz de bombear las drogas citotóxicas fuera de la célula (con muy mal pronóstico, derivada del gen MDR1).
Las mutaciones en Ras aparecen en el 5-10% de los pacientes con leucemia mieloide aguda (LMA), y muy pocas veces en conjunción con mutaciones FLT3.
Una alta expresión del gen del Meningioma 1 (MN) contribuye a un pronóstico adverso, especialmente en conjunción con otros genes mutantes. MN codifica para una proteína que forma parte del complejo regulador de la transcripción del gen del receptor de ácido retinoico.
Mutaciones en el gen del tumor de Wilms se han descrito en el 10% de los nuevos casos de leucemia mieloide aguda (LMA), aunque la influencia sobre el pronóstico es contradictoria.
CONSIDERACIONES FINALES
La leucemia mieloide aguda se caracteriza a nivel molecular por la presencia de aberraciones cromosómicas y genéticas. Los defectos cromosómicos son particularmente abundantes en los cromosomas 5, 7 y 8. Los genes que con mayor frecuencia aparecen mutados en este enfermedad son NPM1, FLT3, CEBPA, RAS, KIT, MLL, p53 y otros.
Aunque FLT3 aparezca mutado y aporte sobre un pronóstico negativo, ninguna mutación por sí sola es capaz de convertir la célula en leucémica, sino que se requieren la acumulación de varios cambios.
Generalmente se puede correlacionar un pronóstico con las mutaciones observadas.
BIBLIOGRAFÍA
1. Appelbaum FR, Gundacker H, Head DR, et al. Age and acutemyeloid leukemia. Blood. 2006;107:3481–3485.
2. Becker H, Marcucci G, Maharry K, et al. Favorable prognostic impact of NPM1 mutations in older patients with cytogenetically normal de novo acute myeloid leukemia and associated gene- and microRNAexpression signatures: a Cancer and Leukemia Group B study. J Clin Oncol. 2010;28:596–604.
3. Beghini A., Ripamonti C., Cairoli R., Cazzaniga G., Colaprieto P., Elice F., Nadali G., Grillo G.,Haas O., Biondi A., Morra E., Larizza L., KIT activating mutations: incidence in adult and pediatric acute myeloid leukemia, and identification of an internal tandem duplication. Haematologica. 2004 Aug;89(8):920-5.
4. Byrd JC, Mrozek K, Dodge RK, et al. Pretreatment cytogenetic abnormalities are predictive of induction success, cumulative incidence of relapse, and overall survival in adult patients with de novo acute myeloid leukemia: results from Cancer and Leukemia Group B (CALGB 8461). Blood. 2002;100:4325– 4336.
5. Cairoli R, Beghini A, Grillo G, Nadali G, Elice F, Ripamonti CB, Colapietro P, Nichelatti M, Pezzetti L, Lunghi M, Cuneo A, Viola A, Ferrara F, Lazzarino M, Rodeghiero F, Pizzolo G, Larizza L, Morra E. Prognostic impact of c-KIT mutations in core binding factor leukemias: an Italian retrospective study. Blood. 2006 May 1;107(9):3463-8.
6. Fröhling S., Schlenk R.F., Stolze I., Bihlmayr J., Benner A, Kreitmeier S., Tobis K., Döhner H., Döhner D., CEBPA Mutations in Younger Adults With Acute Myeloid Leukemia and Normal Cytogenetics: Prognostic Relevance and Analysis of Cooperating Mutations. J Clin Oncol. 2004; 22(4): 624-633.
7. Gale RE, Green C, Allen C, et al. The impact of FLT3 internal tandem duplication mutant level, number, size, and interaction with NPM1 mutations in a large cohort of young adult patients with acute myeloid leukemia. Blood. 2008;111:2776–2784.
8. Hitoshi K., Tomoki N., Yasuyuki N., Shohei Y., Saburo M., Shuichi M., Norio A., Kazutaka K., Itsuro J., Chihiro S., Hideki A., Kenji S., Hakumei O., Toshiko M., Eijiro O., Hidehiko S., Ryuzo O., Ryuzo U. Prognostic Implication of FLT3 and N-RAS Gene Mutations in Acute Myeloid Leukemia. Blood, 1999, 93(9): 3074-3080.
9. Kumar V.; Abbas A.; Fausto N.; Mitchell R.N.; Robbins Patología Humana. Elsevier España, 2008, 179-230.
10. Marcucci G, Maharry K, Wu YZ, et al. IDH1 and IDH2 gene mutations identify novel molecular subsets within de novo cytogenetically normal acute myeloid leukemia: a Cancer and Leukemia Group B Study. J Clin Oncol. 2010;28:2348–2355.
11. Paschka P, Marcucci G, Ruppert A, et al. Adverse prognostic significance of KIT mutations in adult acute myeloid leukemia with (inv 16) and t(8;21): a Cancer and Leukemia Group B study. J Clin Oncol. 2006;24:3904–3911.
12. Perrotti D., Marcucci G., Caligiuri M.A., Loss of C/EBPα and Favorable Prognosis of Acute Myeloid Leukemias: A Biological Paradox Journal of Clinical Oncology, Vol 22, No 4 (February 15), 2004: pp. 582-584.
13. Takahashi S., Downstream molecular pathways of FLT3 in the pathogenesis of acute myeloid leukemia: biology and therapeutic implications. J. Hematol. Oncol. 2011, 4:13.
14. Tamai H., Inokuchi K., 11q23 /Acute Leukemia: Update of Clinical Aspect. J Clin Exp. 2010, 50(2): 91-98.